Uso de herramientas de genómica para la mejora de cultivos / Jerome Grimplet

Jerome Grimplet
Unidad de Hortofruticultura
![]() Centro de Investigación y Tecnología Agroalimentaria de Aragón (CITA)
Centro de Investigación y Tecnología Agroalimentaria de Aragón (CITA)
Fundación Agencia Aragonesa para la Investigación y el Desarrollo (ARAID)
Instituto Agroalimentario de Aragón (IA2)
En varias áreas de la innovación, las herramientas de biotecnología están alcanzando la madurez necesaria para crear una revolución en el mundo agrario. En el campo de la mejora hay que decir que las consecuencias de masificar la secuenciación de genomas, por ejemplo, no están claras para el público y producen desconfianza. Existe actualmente capacidad para saber todo acerca de la genética de un individuo. Este ejercicio se puede hacer sencillamente en unos minutos y un con coste relativamente asequible. Esta tecnología es conocida desde hace años y el genoma humano fue secuenciado en 2001, pero ha bajado significativamente el coste desde entonces y la tecnología se ha miniaturizado hasta poder ser portátil en el campo. ¿Qué consecuencias va a tener en la agricultura y cómo nos va a ayudar a enfrentarnos a los retos climáticos y ambientales que se avecinan?
El mayor impacto sobre la mejora de plantas se va notar en las posibilidades de basar la selección, no tanto en el fenotipo, mediante la selección de individuos con caracteres físico químico de interés como se ha hecho históricamente, sino utilizando el genotipo con marcadores moleculares. Mi colega Celia Cantin recientemente publicó un artículo en esta sección sobre sus utilizaciones (http://opiniones-y-experiencias.chil.me/post/la-utilizacion-de-marcadores-moleculares-en-los-programas-de-mejora-de-melocoton-242619). La ventaja es que es posible conocer muy pronto el genotipo en el desarrollo de la planta, en comparación a esperar a que una planta llegue a su madurez. Así hablamos de herramientas que permite una selección más rápida del mismo material que hubiera sido seleccionado en mejora clásica, sin que se produzca la introducción de genes exógenos. El interés económico es alto, la selección es más precoz ya que se hace directamente en embriones o en plántulas, lo que permite reducir el tiempo de generación de nuevas variedades y los costes de mantenimiento, de manera drástica. Además, más allá del debate sobre la ética de los organismos genéticamente modificados, nos están esperando desafíos con peligros directos y conocidos que tenemos que solucionar. El futuro de las variedades pasa por aumentar la rapidez de su mejora genética para adaptarlas a las nuevas condiciones de producción, cada día más incierta con el cambio climático, a las necesidades de un mercado más volátil y a aumentar su tolerancia a patógenos y plagas en un mundo global.
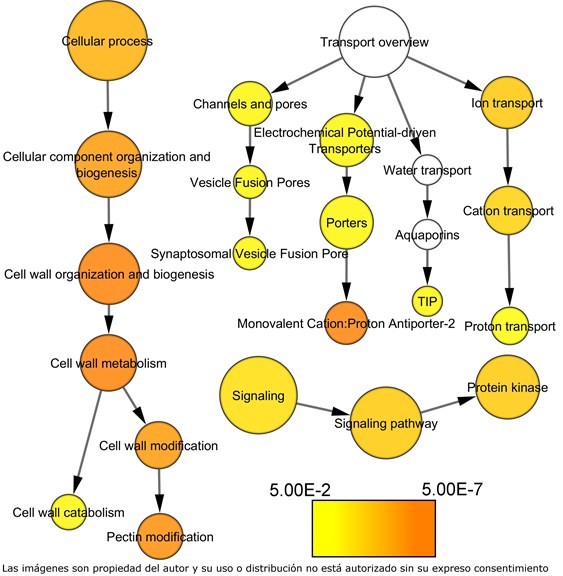
Figura 1 Categorías funcionales de genes más abundante en clones de vid más compactos
La mejora asistida por marcadores no es nueva y es particularmente útil para descartar, en particular, individuos con un fenotipo letal llevando los marcadores asociados/relacionados. En el futuro el mejorador tendrá acceso a un abanico, casi infinito, de estos marcadores de cualquier carácter.
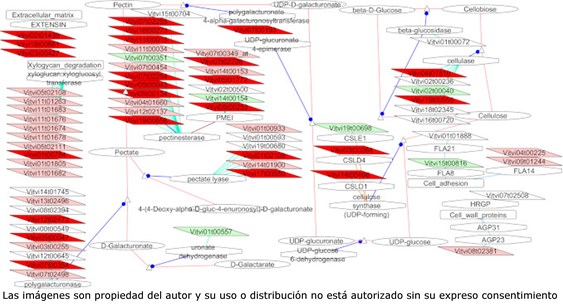
Figura 2: Genes más abundantes en clones de vid más compactos relacionados a la modificación de la pared celular.
La parte más grande del trabajo que queda, no es tanto producir datos sino relacionar los marcadores con caracteres de fenotipo. Para hacer frente a estos retos, la comunidad investigadora debe generar e interpretar grandes conjuntos de datos heterogéneos que describen genotipos y fenotipos así como la interactuación entre ellos y con el medio ambiente. Cuando estos datos son generados y estudiados exhaustivamente, se habla de “ómica”, un término que agrupa áreas de investigación específica como genómica, epigenómica, transcriptómica, proteómica, metabolómica o fenómica. Así, esas áreas de investigación requieren la utilización de herramientas bioinformáticas para tratar grandes cantidades de datos. La integración de estos conjuntos de datos multiómicos consiste a entender las interacciones entre moléculas responsables de los caracteres del fenotipo. Por esto es importante agrupar el conocimiento generado por la comunidad científica dentro de modelos universales. Establecer la relación entre variaciones molecular (estructurales o cuantitativas) y variación de fenotipo reduce la brecha entre la generación de datos y la capacidad de plantear una hipótesis sobre fenotipos de interés a partir del genoma y entonces identificar nuevos marcadores moleculares.
La Unidad de Hortofruticultura del Centro de Investigación y Tecnología Agroalimentaria de Aragón (CITA) tiene la ambición de producir variedades adaptadas al futuro del mercado. Por eso, está desarrollando estrategias que requieren el uso de estas herramientas ómicas en las partes iniciales de los programas de mejora (floración, vigor, adaptación al suelo, producción frutal, épocas de maduración, resistencias…) para definir los marcadores de interés. Por esto necesitamos desarrollar nuevas herramientas de análisis de datos en nuestros campos de interés, por ejemplo, para visualizar la expresión de los genes expresados en individuos con fenotipo de interés (Visualización a nivel categoría funcionales: Figura 1. Visualización al nivel de genes individuales: Figura 2). Además, esto implica una coordinación entre investigadores para que los datos sean compatibles entre ellos y, por lo tanto, se puedan compartir. Por eso, el CITA está participando en el Proyecto Europeo COST “Data integration to maximise the power of omics for grapevine improvement” con el objetivo de tener homogeneidad en los formatos de datos, formación de los investigadores en bioinformática en usar herramientas así como en buenas prácticas de análisis y publicaciones.
